x <- rnorm(n = 100, mean = 10)
#> Calcula a média geométrica
exp(mean(log(x)))[1] 10.221Vinicius Oike
September 15, 2023
A partir da versão 4.1.0, o R passou a oferecer o operador |> chamado de pipe (literalmente, cano)1. Este operador foi fortemente inspirado no operador homônimo %>% do popular pacote magrittr. Neste post, explico como utilizar o pipe nativo e como ele difere do pipe do magrittr.
O operador pipe (em ambos os casos), essencialmente, ordena uma função composta.
Lembrando um pouco sobre funções compostas: a expressão abaixo mostra a aplicação de três funções onde primeiro aplica-se a função f sobre x, depois a função g e, por fim, a função h. Lê-se a função de dentro para fora.
\[ h(g(f(x))) = \dots \]
Para tornar o exemplo mais concreto considere o exemplo abaixo onde calcula-se a média geométrica de uma sequência de números aleatórios.
A média geométrica é dada pela expressão:
\[ \overline{x} = (\prod_{i = 1}^{n}x_{i})^{\frac{1}{n}} = \text{exp}(\frac{1}{n}\sum_{i = 1}^{n}\text{log}(x_{i})) \]
Usando a mesma notação acima, aplica-se primeiro a função log (f), depois a função mean (g) e, por fim, a função exp (h). Usando o operador pipe, pode-se reescrever a expressão da seguinte forma.
Note que o resultado da função vai sendo “carregado” da esquerda para a direita sucessivamente. Para muitos usuários, a segunda sintaxe é mais intuitiva e/ou fácil de ler. No segundo código a ordem em que o nome das funções aparecem coincide com a ordem da sua aplicação.
Por fim, note que o uso de várias funções numa mesma linha de código também nos poupa de ter de criar objetos intermediários como no exemplo abaixo.
[1] 10.221Os exemplos acima funcionaram sem problemas porque usou-se o operador pipe para “abrir” uma função composta. O argumento de cada função subsequente é o resultado da função antecedente: funciona como uma linha de montagem, em que cada nova etapa soma-se ao resultado da etapa anterior.
Quando o resultado da função anterior não vai diretamente no primeiro argumento da função subsequente, precisa-se usar o operador _ (underline/underscore)2. Este operador serve como um placeholder: indica onde que o resultado da etapa anterior deve entrar. No exemplo abaixo, uso o placeholder para colocar a base de dados filtrada no argumento data dentro da função lm.
Por fim, temos o caso das funções anônimas3. Uma função anônima é simplesmente uma função sem nome que é chamada uma única vez. Infelizmente, a sintaxe de um pipe com uma função anônima é bastante carregada.
O exemplo repete o código acima, mas agora usa uma função anônima para pegar o R2 ajustado da regressão.
Imagine agora que se quer calcular o erro absoluto médio de uma regressão. Lembre-se que o EAM é dado por
\[ \text{EAM} = \frac{1}{N}\sum_{i = 1}^{N}|e_{i}| \]
onde \(e_{i}\) é o resíduo da regressão. O código abaixo mostra como fazer isto usando pipes.
#> Estima uma regressão qualquer
fit <- lm(mpg ~ wt, data = mtcars)
#> Calcula o erro absoluto médio
fit |> residuals() |> abs() |> mean()[1] 2.340642Note, contudo, que a situação fica um pouco mais complicada no caso em que se quer calcular a raiz do erro quadrado médio.
\[ \text{REQM} = \sqrt{\frac{1}{N}\sum_{i = 1}^{N}(e_{i})^2} \]
Na sintaxe convencional temos
O problema é que a exponenciação acontece via um operador e não uma função. Nenhum dos exemplos abaixo funciona.
Error: <text>:1:23: unexpected '^'
1: fit |> residuals() |> ^
^Para chegar no mesmo resultado, novamente precisa-se usar uma sintaxe bastante esotérica que envolve passar o resultado de residuals para uma função anônima.
Assim, apesar de muito útil, o operador pipe tem suas limitações. O operador sempre espera encontrar uma função à sua direita; a única maneira de seguir |> com um operador é criando uma função anônima, cuja sintaxe é um pouco carregada. Pode-se resumir os principais fatos sobre o operador pipe:
x |> f |> g o operador |> aplica a função f sobre o objeto x usando x como argumento de f. Depois, aplica a função g sobre o resultado de f(x). Isto é equivalente a g(f(x)).x |> f(y = 2, data = _).x |> (\(y) {funcao})().O uso mais comum de pipes é junto com funções do tidyverse, que foram desenvolvidas com este intuito.
As funções do tidyverse (quase) sempre recebem um data.frame como primeiro argumento; isto facilita a construção de código usando pipe, pois basta encadear as funções em sequência.
A leitura do código fica mais “gramatical”: pegue o objeto mtcars filtre as linhas onde wt > 2 depois agrupe pela variável cyl e, por fim, tire uma média de mpg.
Pode-se terminar um pipe com uma chamada para um plot em ggplot2 para uma rápida visualização dos resultados
mtcars |>
filter(wt > 2) |>
group_by(cyl) |>
summarise(avg = mean(mpg)) |>
ggplot(aes(x = as.factor(cyl), y = avg)) +
geom_col()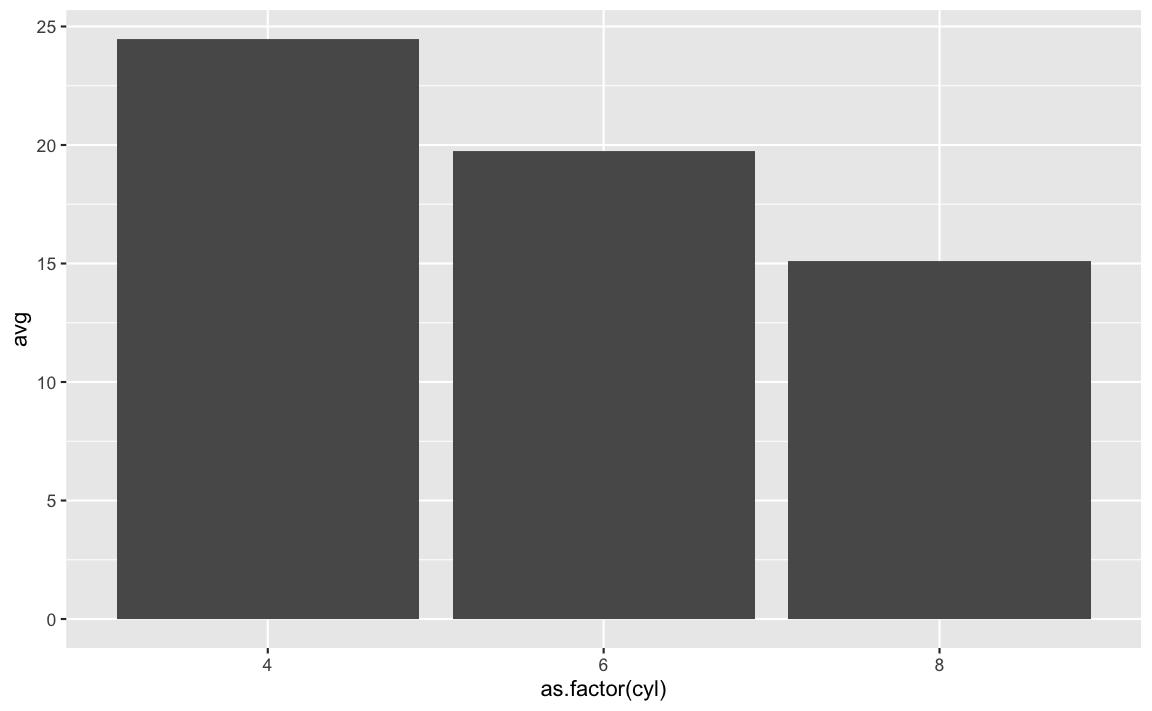
Não se recomenda fazer longas sequências de pipes, pois o código pode acabar muito confuso para quem está lendo. O exemplo abaixo mostra justamente isto.
library(realestatebr)
abecip <- get_abecip_indicators(cached = TRUE)
abecip$units |>
pivot_longer(-date) |>
mutate(yq = lubridate::floor_date(date, unit = "quarter")) |>
group_by(yq, name) |>
summarise(total = sum(value)) |>
separate(name, into = c("category", "type")) |>
filter(category == "units", type != "total") |>
ggplot(aes(x = yq, y = total, fill = type)) +
geom_area() +
scale_fill_brewer() +
theme_light() +
theme(legend.position = "top")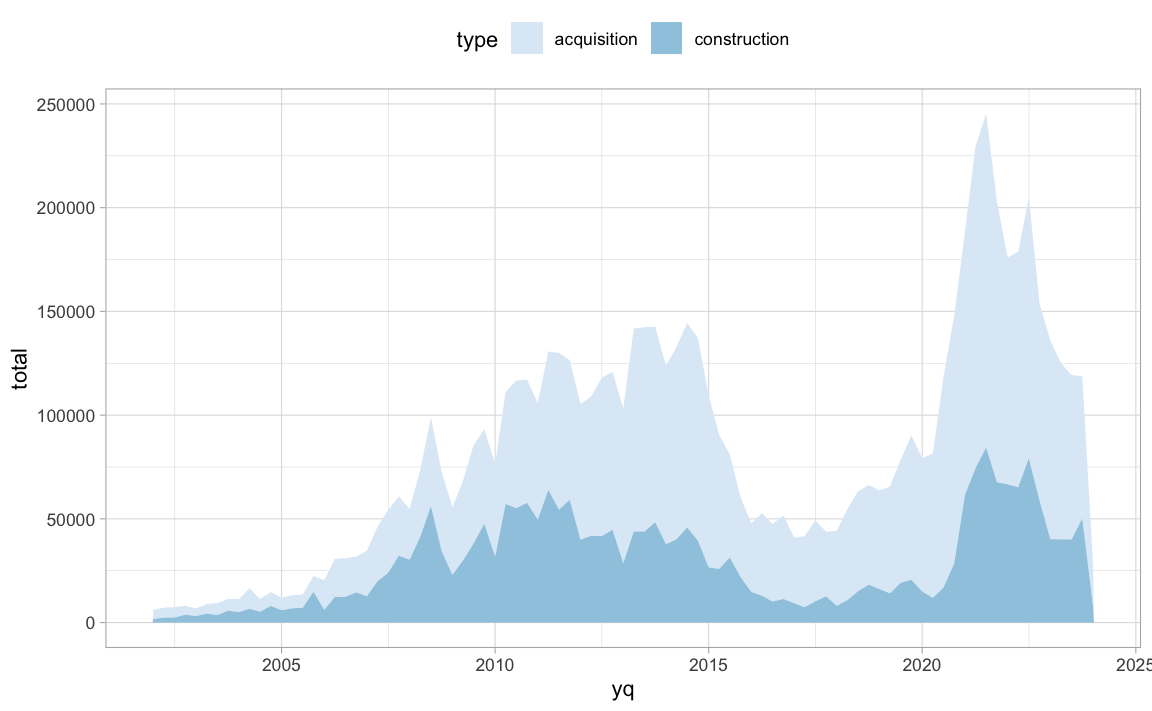
É recomendável quebrar o código acima em passos distintos. Além de ficar mais organizado, pode-se salvar objetos úteis como a tabela agrupada por trimestre, antes de se aplicar o filtro de unidades. A tabela final também fica salva num objeto, permitindo que se faça outros gráficos e análises com estes dados.
units <- abecip$units
#> Converte em long e agrega os dados por trimestre
tab_quarter <- units |>
pivot_longer(-date) |>
mutate(yq = lubridate::floor_date(date, unit = "quarter")) |>
group_by(yq, name) |>
summarise(total = sum(value)) |>
separate(name, into = c("category", "type"))
#> Filtra apenas dados de unidades e retira o 'total'
tab_units <- tab_quarter |>
filter(category == "units", type != "total")
#> Faz o gráfico
ggplot(tab_units, aes(x = yq, y = total, fill = type)) +
geom_area() +
scale_fill_brewer() +
theme_light() +
theme(legend.position = "top")O pacote sf também funciona bem com pipes pois há vários casos em que se quer aplicar múltiplas funções num mesmo objeto.
O exemplo abaixo é emprestado do pacote censobr e mostra como combinar a manipulação de dados do dplyr com objetos espaciais manipulados via sf.
library(censobr)
library(geobr)
library(sf)
library(mapview)
# Importa alguns dados do Censo IBGE 2010
pop <- read_population(
year = 2010,
columns = c("code_weighting", "abbrev_state", "V0010")
)
# Calcula a população total das áreas de ponderação no Rio de Janeiro
df <- pop |>
filter(abbrev_state == "RJ") |>
group_by(code_weighting) |>
summarise(total_pop = sum(V0010)) |>
collect()
# Import o shape das áreas de ponderação do Censo
areas <- read_weighting_area(3304557, showProgress = FALSE)
areas |>
# Converte o CRS da geometria
st_transform(crs = 4326) |>
# "Limpa" as geometrias
st_make_valid() |>
# Junta com os dados do Censo
left_join(df, by = "code_weighting") |>
# Visualiza os dados num mapa interativo
mapview(zcol = "total_pop")O guia de estilo do tidyverse propõe algumas orientações gerais sobre o uso de pipes. Abaixo eu reutilizao vários exemplos do guia.
Primeiro, deve-se evitar de fazer um pipe long numa mesma linha. Esta recomendação visa melhorar a leitura do código. Também se recomenda deixar um espaço em branco antes do |>.
# Bom
iris |>
group_by(Species) |>
summarise(across(where(is.numeric), mean)) |>
ungroup() |>
pivot_longer(-Species, names_to = "measure") |>
arrange(value)
# Ruim
iris |> group_by(Species)|>
summarise(across(where(is.numeric), mean))|>ungroup() |>
pivot_longer(-Species, names_to = "measure") |> arrange(value)# A tibble: 12 × 3
Species measure value
<fct> <chr> <dbl>
1 setosa Petal.Width 0.246
2 versicolor Petal.Width 1.33
3 setosa Petal.Length 1.46
4 virginica Petal.Width 2.03
5 versicolor Sepal.Width 2.77
6 virginica Sepal.Width 2.97
7 setosa Sepal.Width 3.43
8 versicolor Petal.Length 4.26
9 setosa Sepal.Length 5.01
10 virginica Petal.Length 5.55
11 versicolor Sepal.Length 5.94
12 virginica Sepal.Length 6.59 No caso de funções longas, deve-se quebrar/indentar o código.
#> Bom
iris |>
group_by(Species) |>
summarise(
Sepal.Length = mean(Sepal.Length),
Sepal.Width = mean(Sepal.Width),
Species = n_distinct(Species)
)
#> Bom
iris |>
group_by(Species) |>
summarise(Sepal.Length = mean(Sepal.Length),
Sepal.Width = mean(Sepal.Width),
Species = n_distinct(Species))
#> Ruim
iris |>
group_by(Species) |>
summarise(Sepal.Length = mean(Sepal.Length), Sepal.Width = mean(Sepal.Width), Species = n_distinct(Species))Este aqui é um gatilho pessoal; tento evitar ao máximo usar um pipe com uma única função. Pipes são úteis quando se aplica uma sequência de funções a um mesmo objeto; faz pouco sentido usar um pipe quando se aplica somente uma função.
É possível utilizar pipes dentro de funções para fazer pequenas transformações. Pessoalmente, acho que isto deixa o código confuso; quase sempre vale a pena criar um objeto intermediário ao invés de usar o pipe.
O código abaixo, por exemplo, usa a base economics_long, que reúne um conjunto de séries de tempo, e indexa elas na primeira observação. Assim o primeiro valor de cada série tem valor igual a 100 e os valores subsequentes são “proporcionais” a este valor inicial.
Para chegar neste cálculo eu uso um pipe, dentro de uma sequência de pipes.
A versão alternativa do código cria um objeto intermediário com os valores iniciais de cada uma das séries.
Faço um gráfico do resultado final para tornar evidente o que está acontecendo.
Como comentei acima, o pipe nativo do R foi inspirado no pipe do pacote magrittr, um dos mais populares pacotes criados. Há algumas pequenas diferenças entre o %>% e o |>.
Primeiro, em termos de eficiência, o |> é levemente mais rápido, mas a diferença é praticamente imperceptível.
Unit: microseconds
expr min lq mean median uq
{ mean(rnorm(n = 1000)) } 28.536 29.0075 30.17026 29.315 30.094
{ rnorm(n = 1000) %>% mean() } 29.438 29.9300 30.82503 30.299 31.078
max neval cld
58.015 100 a
52.726 100 aA principal diferença entre os dois é o uso do placeholder. O operador placeholder do magrittr é o ponto . e é mais versátil que o _. No exemplo abaixo, eu consigo colocar a tabela filtrada dentro da função rownames durante o pipe.
Isto não funciona com o pipe nativo.
Isto pode ser bastante conveniente quando se lida com objetos espaciais. Imagine o caso em que se tem um objeto criado com sf que guarda polígonos e se quer encontrar as coordenadas dos centróides destes polígonos.
Também consegue-se usar o . junto com operadores e não apenas com funções. O código abaixo calcula a raiz do erro quadrado médio dos resíduos da regressão. Como vimos acima, isto não é possível com o pipe nativo e exigiria uma função anônima.
O . pode ser seguido de outros operadores como o $4. Assim pode-se extrair uma coluna específica depois de filtrar uma tabela.
O . também pode ser fornecido para múltiplos argumentos como no exemplo abaixo.
[1] mpg cyl disp hp drat wt qsec vs am gear carb
<0 rows> (or 0-length row.names)Por fim, o pipe nativo também é inútil quando se usa o pacote data.table. Isto acontece por causa da maneira como o data.table encadeia as suas operações.
Na sintaxe do data.table é possível encadear as operação naturalmente usando [.
# Calcula a média de 'mpg' por 'cyl' e depois ordena segundo 'cyl'
dtmtcars[, .(mpg = mean(mpg)), by = cyl][order(cyl)] cyl mpg
<num> <num>
1: 4 26.66364
2: 6 19.74286
3: 8 15.10000Muitos, contudo, preferem usar o pipe %>% para encadear as operações, já que o código fica mais legível (especialmente em operações mais longas).
cyl mpg
<num> <num>
1: 4 26.66364
2: 6 19.74286
3: 8 15.10000Novamente, como o placeholder do pipe nativo espera sempre receber uma função, o código abaixo não funciona.
Error: pipe placeholder can only be used as a named argumentA solução, como vimos acima, seria criar uma função anônima. Neste caso, também seria possível disfarçar o [ como uma função (por favor, não faça isto).
O pipe é um operador simples que serve para deixar o código mais limpo. Sempre é uma boa escolha usar pipes quando se quer aplicar múltiplas funções num mesmo objeto. O custo do |> é mínimo em termos de eficiência; além disso, ele diminui a necessidade de criar objetos intermediários o que poupa memória do sistema.
O pipe nativo é embutido no R a partir da versão 4.1.0 enquanto o %>% exige library(magrittr). Vale notar que muitos pacotes carregam o %>% automaticamente. De fato, pacotes como dplyr, leaflet, mapview, rvest, gt, flextable e tantos outros são praticamente inutilizáveis sem pipes5.
Antes de pensar em usar o pipe nativo, vale a pena reforçar que ele foi criado recentemente. Isto significa que qualquer código ou pacote desenvolvido com uso de pipes vai exigir a versão 4.1.0 ou superior, o que pode exigir que o usuário atualize a sua versão do R. Além disso, algumas funcionalidades do pipe nativo mudaram nas versões 4.2 e 4.3 o que pode complicar ainda mais o uso do pipe. Em contrapartida, o pipe do magrittr é bastante estável e seu comportamento é consistente há muitos anos.
Pessoalmente, tento utilizar o |> nos posts do blog, mas sem nunca utilizar o _. Quando preciso de um placeholder utilizo o %>%, mas evito usá-lo com operadores como .^2.
Para a lista completa de mudanças veja News and Notes.↩︎
Tecnicamente, o placeholder foi apenas introduzido na versão 4.2.0 como uma melhoria em relação ao pipe nativo implementado anteriormente. “In a forward pipe |> expression it is now possible to use a named argument with the placeholder _ in the rhs call to specify where the lhs is to be inserted. The placeholder can only appear once on the rhs.”. Link original.↩︎
A notação abaixo de função anônima, usando \(x), também foi introduzida na versão 4.1.0 do R. Antigamente, para se definir uma função era necessário usar function(x).↩︎
Vale notar que este comportamento foi introduzido também no placeholder do pipe nativo a partir da versão 4.3.0 do R. Contudo, este comportamento ainda está em fase experimental. “As an experimental feature the placeholder _ can now also be used in the rhs of a forward pipe |> expression as the first argument in an extraction call, such as _$coef.”. Link Original.↩︎
Uma quantidade enorme de pacotes utiliza o magrittr como dependência. Veja a página do CRAN.↩︎